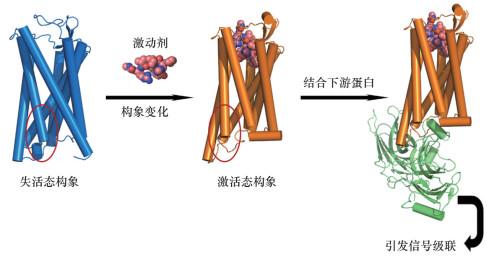
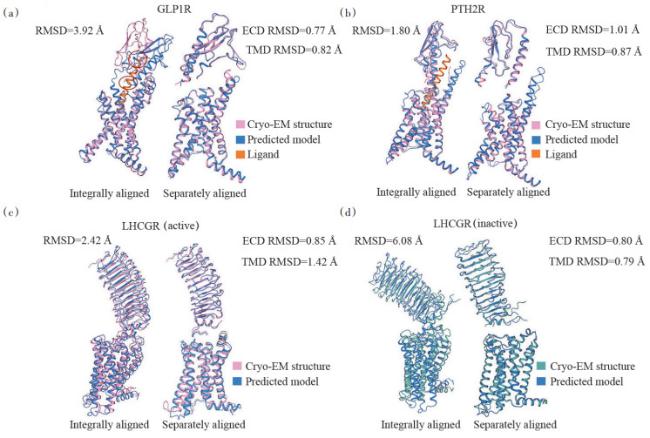
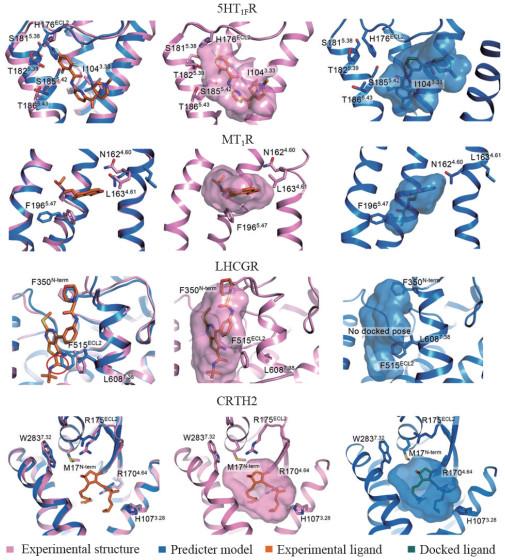
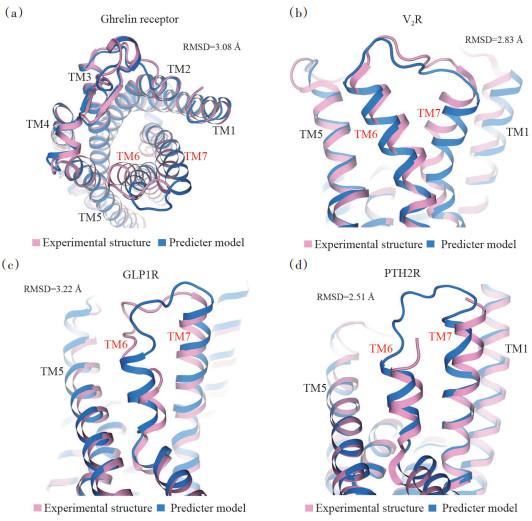
1 AlphaFold与结构生物学
表1 3代AlphaFold的核心差异对比 |
| 版本 | 架构特点 | 关键技术 | 应用和性能 |
| AlphaFold1 | 卷积神经网络 | 序列对齐信息、物理化学约束 | 精度有限,局限于简单蛋白 |
| AlphaFold2 | Transformer+图神经网络 | Evoformer、结构模块、注意力机制 | 精度接近实验水平,适用于广泛单体蛋白预测 |
| AlphaFold3 | Pairformer+扩散模型 | 蛋白质与DNA、RNA、配体、离子等相互作用预测,翻译后修饰影响 | 提高复合物预测精度,助力药物设计和生物学研究 |